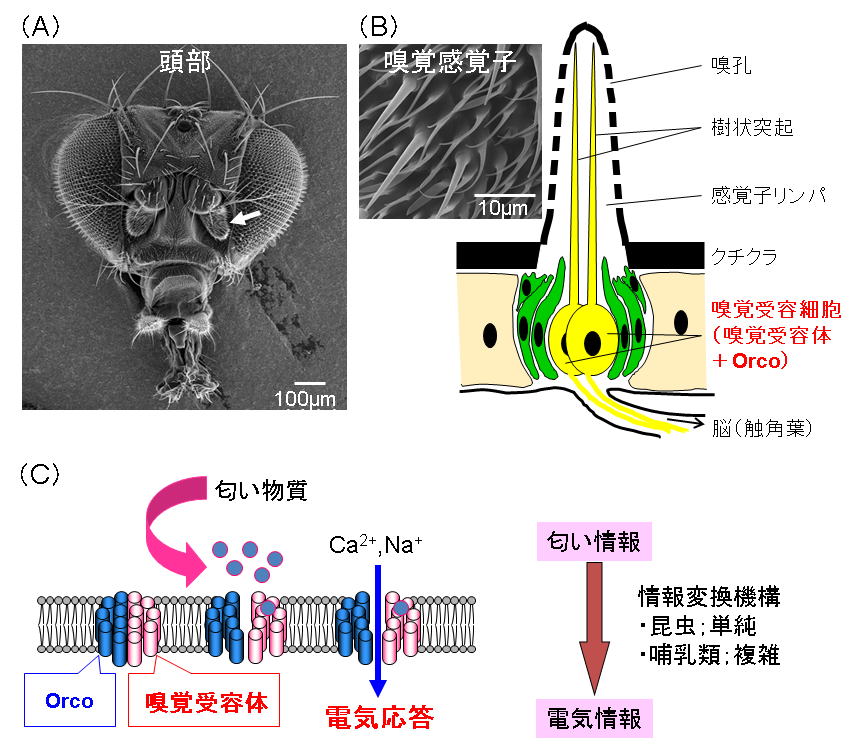
図1 生物の嗅覚受容器官の構造と分子メカニズム
(A)昆虫頭部の走査電子顕微鏡写真。矢印は触角を示す。(B)嗅覚感覚子の電子顕微鏡写真と模式図。匂い物質は嗅孔から入り、感覚子リンパ中の可溶性タンパク質で輸送され、樹状突起膜上に存在する嗅覚受容体に到達する。その後、匂い情報は嗅覚受容細胞で電気情報へと変換され、脳(触角葉)に伝達される。(C)昆虫生体における嗅覚受容の分子メカニズム。嗅覚受容体は匂い物質を受容すると、細胞外から細胞内へ陽イオン(Ca2+、Na+)を流入する。二次情報伝達物質を介した複雑な情報変換機構を採用する哺乳類の嗅覚受容体と比べ、昆虫の嗅覚受容体は単純な情報変換機構により細胞内へシグナルを伝える。
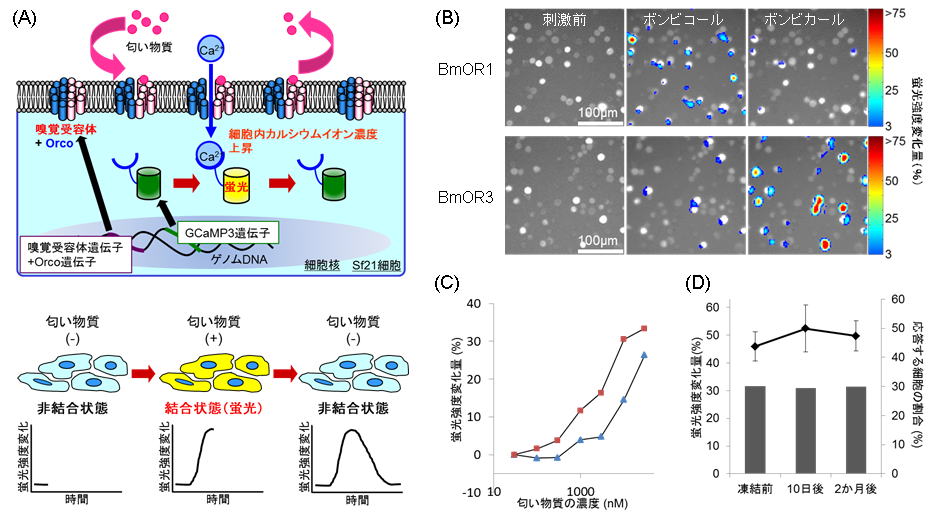
図2 センサ細胞の匂い検出の模式図と性能評価
(A)センサ細胞の応答メカニズム。昆虫の嗅覚受容体遺伝子、Orco遺伝子及びカルシウム感受性蛍光タンパク質(GCaMP3)遺伝子をゲノムDNAに組み込んだSf21細胞系統を樹立した。非結合状態の嗅覚受容体に、匂い物質が結合すると、イオンチャネルが開く。これにより、細胞外からカルシウムイオンが流入し、GCaMP3と結合することで、細胞の蛍光強度が上昇する。嗅覚受容体が匂い分子を解離し非結合状態になると、イオンチャネルが閉じ、蛍光が基底状態へと戻る。(B)BmOR1系統とBmOR3系統の匂い選択性。各細胞系統にそれぞれ10µMのボンビコール、10µMのボンビカールを添加した。BmOR1系統はボンビコールに特異的に、BmOR3系統はボンビカールに特異的に蛍光応答を示した。細胞の蛍光強度変化量は疑似カラーで表した。カラーバーは⊿F(蛍光強度変化量)/F(刺激前の蛍光強度)×100(%)の値を表し、暖色ほど蛍光強度変化量の大きい細胞を表す。(C)BmOR1系統、BmOR3系統におけるボンビコール、ボンビカール刺激に対する応答の濃度依存性。(D)BmOR3系統における長期培養後の匂い検出の安定性。蛍光強度変化量を折れ線グラフ、応答する細胞の割合を棒グラフで示す。10日後、2か月後の細胞系統も凍結前と同じ匂い検出性能を示した。

図3 センサ細胞を導入したチップの構造と応答
(A)マイクロ化学チップ(マイクロ化学技研株式会社製)の構造。チップはガラス及びアルミニウムフレームからなる。注入口から細胞や培養液、刺激を注入できる。挿入図は培養部位(白三角印)の拡大写真を示す。(B)チップ中の細胞の蛍光応答。BmOR1系統をチップに導入し、10µMのボンビコールで刺激した。細胞の蛍光強度変化量は疑似カラーで表した。チップ中の細胞から蛍光強度変化を取得できた。
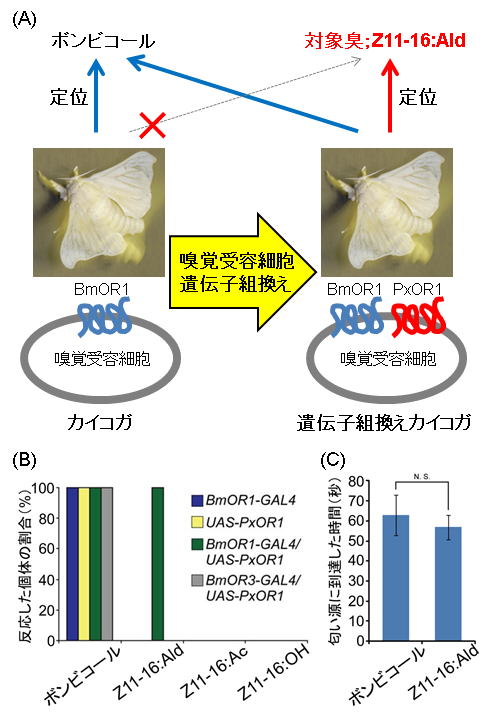
図4 センサ昆虫の概念図と定位行動7)
(A)センサ昆虫の概念図。ボンビコールを受容する嗅覚受容細胞に、対象となるコナガの性フェロモン受容体PxOR1を組み換えたカイコガを作出することで、カイコガが本来応答しないZ11-16:Aldを検出し、発信源に定位するカイコガが作出できる。(B)ボンビコールとコナガ性フェロモン成分に対して行動を発現した個体の割合。PxOR1発現カイコガ(BmOR1-GAL4/UAS-PxOR1)では全個体がZ11-16:Ald(100 ng/filter paper)に反応を示した。BmOR1-GAL4、UAS-PxOR1 はPxOR1 を発現しないバックグランド系統を示す。(C)PxOR1 発現カイコガのボンビコールまたはZ11-16:Ald 匂い源への定位時間の比較。100 ng の各フェロモン成分をしみこませたろ紙をそれぞれ匂い源とした。定位時間に有意差は認められなかった。(参考文献7から改変して引用)
1)Ishida et al., Sens. Actuators B, 33, 115-121, 1996.
2)Hayes et al., IEEE Sensors J, 2, 260-271, 2002.
3)Rau and Rau, Trans. Acad. Sci., 26, 83-221, 1929.
4)Arshak et al., Sensor Review, 24, 181-198, 2004.
5)Glatz and Bailey-Hill, Prog. Neurobiol., 93, 270-296, 2011.
6)Sato et al., Nature, 452, 1002-1006, 2008.
7)Sakurai et al., PLoS Genetics, 7, e1002115, 2011.
8)Misawa et al., Proc. Natl. Acad. Sci. U.S.A., 107, 15340-15344, 2010.
9)Kanzaki et al., Zool Sci, 9, 515-527, 1992.
10)Benton et al., Cell, 136, 149-162, 2009.
11)Hallem and Carlson, Cell, 125, 143-160, 2006.
12)Sakurai et al., Proc. Natl. Acad. Sci. USA, 101, 16653-16658, 2004.
13)Nakagawa et al., Science, 307, 1638-1642, 2005.
14)Mitsuno et al., Eur. J. Neurosci., 28, 893-902, 2008.
15)Kuwana et al., Biosens Bioelectron, 14, 195-202, 1999.